Rodanasa
| Rodanasa | ||||
|---|---|---|---|---|
 | ||||
| Estructuras disponibles | ||||
| PDB | Estructuras enzimáticas RCSB PDB, PDBe, PDBsum | |||
| Identificadores | ||||
| Identificadores externos | Bases de datos de enzimas IntEnz: entrada en IntEnz BRENDA: entrada en BRENDA ExPASy: NiceZime view KEGG: entrada en KEEG PRIAM: perfil PRIAM ExplorEnz: entrada en ExplorEnz MetaCyc: vía metabólica | |||
| Número EC | 2.8.1.1 | |||
| Ortólogos | ||||
| Especies |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
| ||||
[editar datos en Wikidata] | ||||
| Dominio similar a rodanasa | ||
|---|---|---|
| Identificadores | ||
| Símbolo | Rodanasa | |
| Pfam | PF00581 | |
| InterPro | IPR001763 | |
| PROSITE | PDOC00322 | |
| SCOP | 2ora | |
Estructuras PDB disponibles: 1boi 1orb
1rhd 2ora 1dp2 1rhs 1boh 1urh 1h4m 1h4k 1e0c 1tq1 1gn0 1gmx 1qxn 1yt8 1hzm 1c25 1yml 1ymd 2a2k 1ymk 1cwr 1ym9 1cwt 1ys0 1cws 1qb01whb | ||
[editar datos en Wikidata] | ||
La rodanasa es una enzima mitocondrial (EC 2.8.1.1) cuyo nombre sistemático es tiosulfato sulfurotransferasa. Esta enzima detoxifica el cianuro (CN-) convirtiéndolo en tiocianato (SCN-).[1]
Esta reacción ocurre en dos etapas. El diagrama de la derecha muestra la estructura determinada por cristalografía de rayos X de la rodanasa. En el primer paso, el tiosulfato reacciona con el grupo tiol de la cisteína-247 1, para formar un disulfuro 2. En el segundo paso, el disulfuro reacciona con el cianuro para producir tiocianato, mientras que simultáneamente se autoconvierte nuevamente a su estado tiol normal 1.
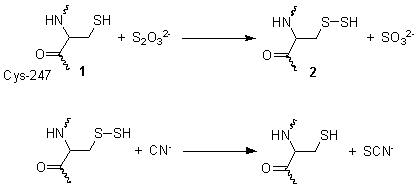
Esta reacción es importante como mecanismo de detoxificación del cianuro, ya que el tiocianato formado es relativamente inocuo. El uso de una solución de tiosulfato como antídoto para el envenenamiento con cianuro se basa en la activación de este ciclo enzimático.
La rodanasa comparte relaciones evolutivas con un gran número de proteínas, entre las que se incluyen:
- Cdc25 dominio catalítico fosfatasa.
- dominios no catalíticos de las fosfatasas MAPK de especificidad dual
- dominios no catalíticos de las fosfatasas MAPK de levadura tipo PTP
- dominios no catalíticos de enzimas de levadura Ubp4, Ubp5, Ubp7
- dominios no catalíticos de enzimas de mamíferos Ubp-Y
- proteína de choque térmico de Drosophila HSP-67BB
- varias proteínas bacterianas de choque por frío y fagos
- proteínas asociadas a la senescencia de plantas
- dominios catalíticos y no catalíticos de rodanasas .
La rodanasa es producto de una duplicación interna. Este dominio se encuentra en forma de copia simple en otras proteínas, incluyendo a fosfatasas y ubiquitin hidrolasas C-terminales.[2]
Proteínas humanas que contienen este dominio
CDC25A; CDC25B; CDC25C; DUSP; DUSP1; DUSP10; DUSP16; DUSP2; DUSP4; DUSP5; DUSP6; DUSP7; KAT; MKP7; MOCS3; MPST; TBCK; TSGA14; TST; USP8;
Referencias
- ↑ Cipollone R, Ascenzi P, Tomao P, Imperi F, Visca P (2008). «Enzymatic detoxification of cyanide: clues from Pseudomonas aeruginosa Rhodanese». J. Mol. Microbiol. Biotechnol. 15 (2-3): 199-211. PMID 18685272. doi:10.1159/000121331.
- ↑ Gliubich F, Gazerro M, Zanotti G, Delbono S, Bombieri G, Berni R (1996). «Active site structural features for chemically modified forms of rhodanese». J. Biol. Chem. 271 (35): 21054-21061. PMID 8702871. doi:10.1074/jbc.271.35.21054.
- F. Gliubich, M. Gazerro, G. Zanotti, S. Delbono, G. Bombieri, R. Berni (1996). «Active Site Structural Features for Chemically Modified Forms of Rhodanese». Journal of Biological Chemistry 271 (35): 21054-21061. PMID 8702871. doi:10.1074/jbc.271.35.21054.
Enlaces externos
- MeSH: Rhodanese (en inglés)
 Datos: Q423139
Datos: Q423139 Multimedia: Rhodanese / Q423139
Multimedia: Rhodanese / Q423139












