| MITF |
|---|
 |
|
| Structures disponibles |
|---|
| PDB | Recherche d'orthologue: PDBe RCSB |
|---|
|
|
| Identifiants |
|---|
| Aliases | MITF |
|---|
| IDs externes | OMIM: 156845 MGI: 104554 HomoloGene: 4892 GeneCards: MITF |
|---|
| Position du gène (Homme) |
|---|
 | | Chr. | Chromosome 3 humain[1] |
|---|
| | Locus | 3p13 | Début | 69,739,456 bp[1] |
|---|
| Fin | 69,968,336 bp[1] |
|---|
|
| Position du gène (Souris) |
|---|
 | | Chr. | Chromosome 6 (souris)[2] |
|---|
| | Locus | 6 D3|6 45.05 cM | Début | 97,784,013 bp[2] |
|---|
| Fin | 97,998,310 bp[2] |
|---|
|
| Expression génétique |
|---|
| Bgee | | Humain | Souris (orthologue) |
|---|
| Fortement exprimé dans | - épithélium pigmentaire rétinien
- Skeletal muscle tissue of biceps brachii
- ventricule droit
- muscle glutéal
- myocarde
- Skeletal muscle tissue of rectus abdominis
- muscle vaste latéral
- myocardium of left ventricle
- muscle deltoïde
- urètre
|
| | Fortement exprimé dans | - glande pinéale
- gastrula
- Caduque basale
- iris
- corps ciliaire
- Epithelium of choroid plexus
- épithélium pigmentaire rétinien
- strie vasculaire
- ventricule droit
- valve auriculo-ventriculaire
|
| | Plus de données d'expression de référence |
|
|---|
| BioGPS | 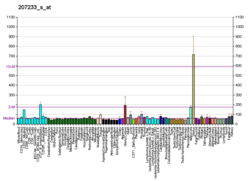 | | Plus de données d'expression de référence |
|
|---|
|
| Gene Ontology |
|---|
| Fonction moléculaire | - liaison ADN
- protein dimerization activity
- DNA-binding transcription factor activity
- liaison protéique
- DNA-binding transcription repressor activity, RNA polymerase II-specific
- RNA polymerase II cis-regulatory region sequence-specific DNA binding
- DNA-binding transcription activator activity, RNA polymerase II-specific
- liaison de chromatine
- transcription factor activity, RNA polymerase II distal enhancer sequence-specific binding
- E-box binding
- DNA-binding transcription factor activity, RNA polymerase II-specific
| | Composant cellulaire | - nucléoplasme
- noyau
- complexe macromoléculaire
| | Processus biologique | - regulation of RNA biosynthetic process
- regulation of transcription by RNA polymerase II
- negative regulation of transcription by RNA polymerase II
- positive regulation of DNA-templated transcription, initiation
- transcription, DNA-templated
- développent d'un organisme multicellulaire
- positive regulation of gene expression
- transcription by RNA polymerase II
- regulation of transcription, DNA-templated
- régulation de l'expression des gènes
- Voie de signalisation Wnt
- différenciation cellulaire
- osteoclast differentiation
- melanocyte differentiation
- regulation of cell population proliferation
- camera-type eye development
- negative regulation of apoptotic process
- pigmentation
- canonical Wnt signaling pathway involved in negative regulation of apoptotic process
- cell fate commitment
- regulation of osteoclast differentiation
- régulation positive de la transcription dépendante de l'ADN
- positive regulation of transcription by RNA polymerase II
- remodelage osseux
- protein-containing complex assembly
- negative regulation of cell migration
| | Sources:Amigo / QuickGO |
|
| Orthologues |
|---|
| Espèces | Homme | Souris |
|---|
| Entrez | | |
|---|
| Ensembl | | |
|---|
| UniProt | | |
|---|
| RefSeq (mRNA) | NM_000248
NM_001184967
NM_001184968
NM_006722
NM_198158
|
|---|
NM_198159
NM_198177
NM_198178
NM_001354604
NM_001354605
NM_001354606
NM_001354607
NM_001354608 |
| |
|---|
NM_001113198
NM_001178049
NM_008601 |
|
|---|
| RefSeq (protéine) | NP_000239
NP_001171896
NP_001171897
NP_006713
NP_937801
|
|---|
NP_937802
NP_937820
NP_937821
NP_001341533
NP_001341534
NP_001341535
NP_001341536
NP_001341537 |
| |
|---|
NP_001106669
NP_001171520
NP_032627 |
|
|---|
| Localisation (UCSC) | Chr 3: 69.74 – 69.97 Mb | Chr 6: 97.78 – 98 Mb |
|---|
| Publication PubMed | [3] | [4] |
|---|
|
| Wikidata |
| Voir/Editer Humain | Voir/Editer Souris |
|

 Portail de la biologie cellulaire et moléculaire
Portail de la biologie cellulaire et moléculaire  Portail de la médecine
Portail de la médecine 















